PCA概述
高维数据劣势
我们很希望有足够多的特征(知识)来保证学习模型的训练效果,尤其在进行图像处理时,高维特征是在所难免的,但是,高维的特征也有几个如下劣势:
学习性能下降,知识越多,吸收知识(输入),并且精通知识(学习)的速度就越慢。
过多的特征难于分辨,你很难第一时间认识某个特征代表的意义。
特征冗余,如下图所示,厘米和英尺就是一对冗余特征,他们本身代表的意义是一样的,并且能够相互转换。
PS这篇实在是没有时间仔细来写了,就简单进行一下叙述,改日再续。。。
PCA的解释一
PCA,Principle Component Analysis,即主成分分析法,是特征降维的最常用手段。PCA 能从冗余特征中提取主要成分,在不太损失模型质量的情况下,提升了模型训练速度。
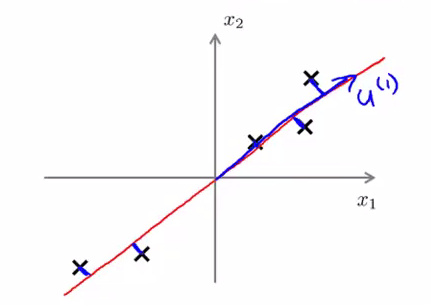
如上图所示,我们将样本到红色向量的距离称作是投影误差(Projection Error)。以二维投影到一维为例,PCA 就是要找寻一条直线,使得各个特征的投影误差足够小,这样才能尽可能的保留原特征具有的信息。
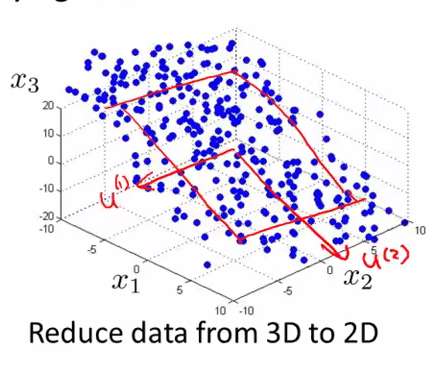
假设我们要将特征从 $n$ 维度降到$k $维:PCA 首先找寻 $k$ 个 $n$ 维向量,然后将特征投影到这些向量构成的$k$维空间,并保证投影误差足够小。下图中,为了将特征维度从三维降低到二位,PCA 就会先找寻两个三维向量
$u^{(1)}$,$u^{(2)}$二者构成了一个二维平面,然后将原来的三维特征投影到该二维平面上:
PCA的解释二
在PCA中,是一个坐标变换的过程,高维数据变到低维数据,数据从原来的坐标系转换到新的坐标系,那么坐标系应当如何选取呢?转换坐标系时,以方差最大的方向作为坐标轴方向,这里的方差最大指数据在该坐标轴的投影的值的方差,因为数据的最大方差给出了数据的最重要的信息。
1.第一个新坐标轴选择的是原始数据中方差最大的方向,
2.第二个新坐标轴选择的是与第一个新坐标轴正交且方差次大的方向
……
3.重复该过程,直至新坐标轴数量足够多。
通过这种方式获得的新的坐标系,大部分方差都包含在前面几个坐标轴中,后面的坐标轴所含的方差远小于前面的。于是,我们可以忽略余下的坐标轴,只保留前面的几个含有绝大部分方差的坐标轴。事实上,这样也就相当于只保留包含绝大部分方差的维度特征,而丢弃了包含方差小的特征维度,也就实现了对数据特征的降维处理。
源代码
1 | ###此段代码来源于《机器学习实战》,为pca.py文件 |
1 | ###此段代码测试文件main.py |